Índice
Un estudio de arqueólogos, bioinformáticos, biólogos y químicos ha revelado los secretos moleculares de los microorganismos del Paleolítico.
El trabajo, que se publica en Science, ha logrado reconstruir los genomas de bacterias desconocidas hasta ahora, a partir del sarro dental de humanos y neandertales.
En la actualidad, el estudio de los productos naturales microbianos se limita en gran medida a las bacterias vivas, pero dado que estos microorganismos han habitado la tierra durante más de tres mil millones de años, existe una enorme diversidad de productos biológicos del pasado con potencial terapéutico –el desarrollo de nuevos medicamentos, por ejemplo–, que siguen siendo desconocidos, dicen los autores.
El equipo científico internacional, liderado desde el Instituto Leibniz de Investigación de Productos Naturales e Infecciones–Hans Knöll Institute y el Instituto Max Planck de Antropología Evolutiva (ambos de Alemania), ha utilizado los patrones genéticos de microorganismos procedentes de la placa dental fosilizada para construir una plataforma biotecnológica que permite ‘revivir’ los productos naturales de las antiguas bacterias.
Genomas bacterianos encerrados en la placa dental
“La idea fundamental de este trabajo es traducir la información genética antigua en un compuesto químico. En términos más generales, nos interesa recuperar información genética de bacterias antiguas para recrear sus funciones arcaicas”, explica a SINC Pierre Stallforth, director de Paleobiotecnología en Leibniz-HKI y uno de los líderes de la investigación.
Pero aclara: “No basamos nuestro estudio únicamente en análisis bioinformáticos, sino que introducimos ADN antiguo en bacterias modernas para ver si pueden producir algo que habrían hecho sus homólogas antiguas. Este enfoque pionero añade una nueva dimensión temporal a la búsqueda de nuevas estructuras químicas”.
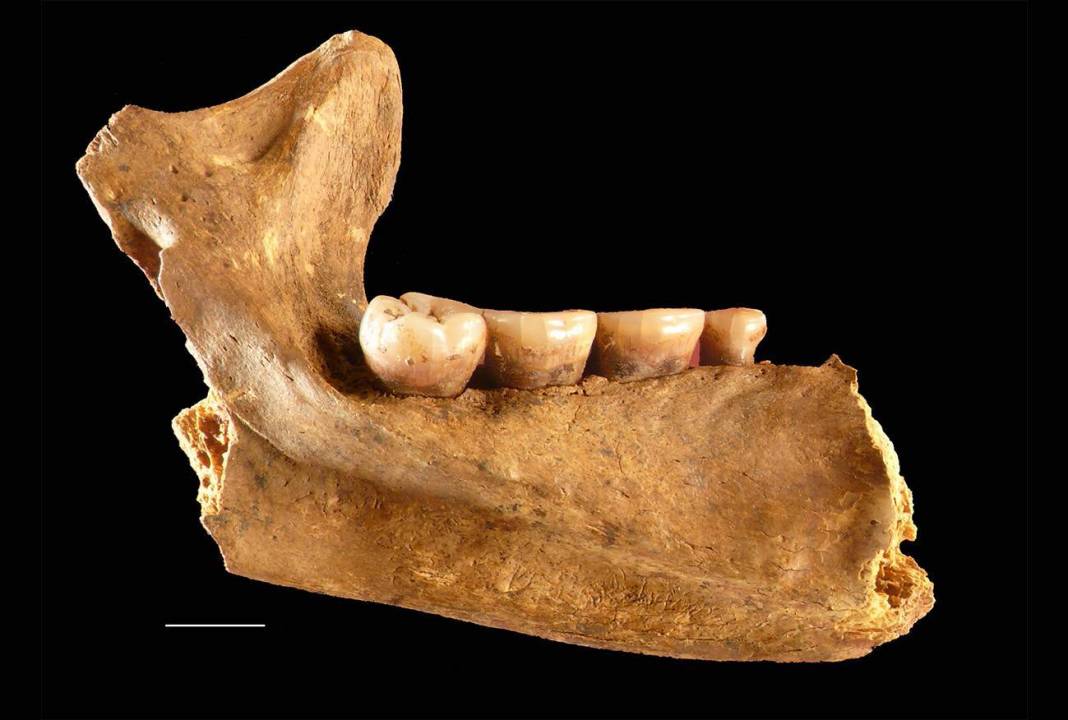
Los autores se centraron en la reconstrucción de genomas bacterianos encerrados en la placa dental de 12 neandertales (de hace unos 102.000–40.000 años), 34 humanos arqueológicos (de entre 30.000 y 150 años atrás) y 18 humanos actuales.
El sarro es la única parte del cuerpo que se fosiliza durante la vida, convirtiendo la placa dental viva en un ‘cementerio’ de bacterias mineralizadas, explican.
El equipo reconstruyó numerosas especies de bacterias orales, así como otras más exóticas cuyos genomas no habían sido descritos antes. Entre ellos, descubrieron una especie bacteriana del Pleistoceno dentro del género Chlorobium, cuyo ADN altamente dañado mostraba las características de una edad ‘avanzada’, y que se encontró en el cálculo dental de siete humanos paleolíticos y neandertales.
Además, los investigadores encontraron que los siete genomas de Chlorobium contenían un grupo de genes biosintéticos de función desconocida.
“El cálculo dental de la ‘Dama Roja’ de El Mirón (Cantabria), de 19.000 años de antigüedad, arrojó un genoma de Chlorobium particularmente bien conservado”, destaca Anan Ibrahim, investigadora Leibniz-HKI y coautora del trabajo.
“Después de haber encontrado estos enigmáticos genes antiguos, queríamos llevarlos al laboratorio para descubrir qué hacían”, subraya.
‘Paleofuranos’
Para ello, utilizaron herramientas de biotecnología molecular sintética que permitieran que las bacterias vivas produjeran las sustancias químicas codificadas por los genes antiguos.
Como resultado, los investigadores descubrieron, además, que estos antiguos grupos de genes biosintéticos producían metabolitos de los que no se había informado anteriormente.
En concreto, productos del ácido 5-alquilfurano-3-carboxílico, a los que han denominado ‘paleofuranos’.
Stallforth recalca a SINC que “es la primera vez que se ha logrado recrear con éxito un producto natural –es decir, un compuesto químico de bajo peso molecular– a partir de ADN bacteriano de hace 100.000 años.
Esto nos permite investigar las funciones de estos compuestos en las antiguas comunidades microbianas para comprender mejor cómo interactuaban las bacterias entre sí o con sus huéspedes”.
Además, agrega, “nuestro método podría ayudar a encontrar nuevos compuestos biológicamente activos que podrían tener actividades antibióticas”.